Typ IV Effektorkomplexe: Komponenten des letzten unbekannten CRISPR-Cas-Typs
Bakterien nutzen CRISPR-Cas-Systeme um sich gegen virale Attacken zu schützen. Auf Grundlage von charakteristischen Cas-Proteinen wurden sechs verschiedene CRISPR-Cas-Typen identifiziert. Fünf dieser sechs Typen wurden bislang genauer untersucht. Wissenschaftler der Max-Planck-Gesellschaft haben nun die Komponenten des letzten unbekannten Typs beschrieben, die unter anderem eine neuartige CRISPR-RNA-Nuklease beinhalten. Die Funktionen der entdeckten Typ IV Effektorkomplexe sind dabei vermutlich nicht der reinen Virenabwehr zugeordnet.
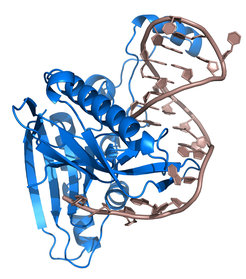
CRISPR-Cas Systeme befinden sich in den Genomen von Bakterien und Archaeen und werden als Immunsysteme genutzt, um fremde DNA zu erkennen und zu zerstören. CRISPR-Abschnitte enthalten dabei Fragmente von viralen Genomen und dienen als zelluläres Gedächtnis vorausgegangener viraler Angriffe. Diese genetische Information wird in CRISPR-RNAs (crRNAs) umgeschrieben, die Cas-Proteine zu ihren Zielsequenzen leiten. Die Koevolution von CRISPR-Cas-Systemen und viralen Anti-CRISPR-Mechanismen führte zu einer Vielzahl an unterschiedlichen CRISPR-Cas-Systemen, von denen sechs Hauptypen und über 30 Subtypen klassifiziert wurden. Viele dieser Systeme wurden im Detail beschrieben und die Effektorkomplexe aus Escherichia coli (Cascade, Typ I) oder das Effektorprotein Cas9 (Typ II) werden weitverbreitet als Werkzeuge zur Editierung von Genomen eingesetzt. Wissenschaftler am Max-Planck-Institut für Terrestrische Mikrobiologie haben nun am Beispiel des Typ IV Systems aus dem Bakterium Aromatoleum aromaticum den Aufbau des letzten verbleibenden CRISPR-Cas Typs aufgeklärt.
In einem kürzlich, in der wissenschaftlichen Fachzeitschrift Nature Microbiology, veröffentlichten Artikel beschreibt Ahsen Özcan, die Erstautorin der Studie, einen neuartigen Weg der Prozessierung von crRNAs, die in Typ IV-spezifische Effektorkomplexe eingebaut werden. Die gemeinschaftliche Arbeit der Forschungsgruppe von Lennart Randau mit den Laboren von Gert Bange, Johann Heider, Thomas Heimerl und Henning Urlaub ermöglichte die Bestimmung von Strukturen der beteiligten Cas-Proteine und ihrer Interaktionen im Komplex. Die Kristallstruktur des Cas-Proteins Csf5 mit einer prozessierten crRNA offenbarte ungewöhnlich kurze 7 nt CRISPR Repeat-Fragmente der 5′-Enden. Elektronenmikroskopie- und Massenspektrometrieuntersuchungen zeigten, dass einzelne crRNA-Moleküle von mehren Csf2-Untereinheiten gebunden werden, die das Rückgrad von sichelförmigen Komplexen bilden, die den Effektorkomplexen von Typ I-Systemen ähneln.
Diese Ähnlichkeiten in Aufbau und Struktur suggerieren, dass auch Typ IV Effektorkomplexe crRNAs nutzen, um virale Ziel-Sequenzen zu erkennen. Allerdings fehlen in diesem Typ Cas-Proteine sowohl zum Einbau von viralen Fragmenten in den CRISPR-Arrays als auch zum Verdau der Zielsequenzen. Daher ist es möglich, dass die Zielerkennung dieser Systeme nicht der Virenabwehr dient und eine mögliche Funktion zur Steuerung der Stabilität von Genomen oder Plasmiden wird postuliert.








