Lichtgesteuerte Injektion im Mikromaßstab
Ein bakterielles Sekretionssystem kann genutzt werden, um Proteine zielgenau in Zellen einschleusen.
Schon seit längerem arbeiten Forscherinnen und Forscher daran, mithilfe eines bakteriellen Injektionsapparates bestimmte Proteine in Eukaryonten-Zellen einzuschleusen. Einem Forscherteam des Max-Planck-Instituts für terrestrische Mikrobiologie unter Leitung von Dr. Andreas Diepold gelang es nun, das Injektionssystem optogenetisch, also mit Licht, zu steuern. Das ermöglicht zukünftig den zielgenauen Einsatz in biotechnologischen oder medizinischen Anwendungen.
Wenn Bakterien wie Salmonella oder Yersinia bei einem Menschen Fieber, Durchfall oder Bauchschmerzen verursachen, sind winzige „Injektionsnadeln“ am Werk: das Typ 3-Sekretionssystem, kurz: T3SS, schießt Virulenzproteine der Bakterien direkt in die eukaryotischen Wirtszellen.
Das bakterielle Injektionssystem als "Trojanisches Pferd"
Da viele Bakterien das T3SS für eine Infektion benötigen, ist es ein zentrales Ziel für mögliche Therapeutika, die seine Funktionsfähigkeit beeinträchtigen und damit Infektionen verhindern sollen. Auf der anderen Seite bietet es die Chance, wie mit einem „trojanischem Pferd“ Proteine nach Wahl in eukaryotische Zellen einzuschleusen. Der Transfer von Proteinen durch T3SS ist schnell and effizient: Eine einzelne Injektisomeneinheit kann in wenigen Sekunden Tausende Effektoren in eine einzelne Wirtszelle übertragen.
So schlagkräftig das T3SS-Injektionssystem jedoch ist, so ungenau arbeitet es - zum Leidwesen der Forschenden. „Sobald T3SS in Kontakt mit einer Wirtszelle kommt, feuert es seine Ladung ab. Das ist ungünstig für Anwendungen der Biotechnologie oder Medizin, bei denen man ja ganz bestimmte Zelltypen im Visier hat, zum Beispiel in der Tumortherapie“, so Andreas Diepold, Arbeitsgruppenleiter am Max-Planck-Institut für terrestrische Mikrobiologie.
Proteininjektion mittels Lichtschalter
Doch nun ist seine Arbeitsgruppe diesem Ziel einen großen Schritt näher gekommen: Es gelang dem Marburger Forscherteam, das T3SS-Injektionssystem vollständig durch Lichteinstrahlung von außen zu steuern (Abb. 1). Damit könnten Proteine zu einem präzisen Zeitpunkt und einem genau bestimmten Ort in eukaryotische Zellen injiziert werden, wie die Autoren in der Fachzeitschrift Nature Communications berichten.
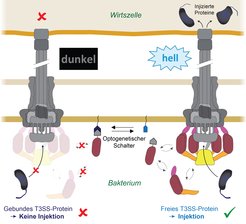
Abb. 1: Funktionsweise des LITESEC-Systems. Im Dunkeln (links) ist eine T3SS-Komponente, die für die Injektion notwendig ist (rot), an der Membran des Bakteriums verankert. Sobald das Bakterium beleuchtet wird (rechts), wird die Komponente freigegeben, das T3SS wird aktiv und Proteine können in die Wirtszelle (oben) injiziert werden.
„Bereits in meiner Zeit als Postdoktorand an der University Oxford entdeckte ich, dass bestimmte Teile des T3SS dynamisch mobil sind“, erklärt Andreas Diepold. „Sie tauschen sich ständig zwischen diesem Apparat und dem Zellinneren aus.“ Diese Erkenntnise verband er mit dem noch jungen Forschungszweig der optogenetischen Steuerung. Das Prinzip dieser Technik: Protein-Interaktionen lassen sich mit einer Schnelligkeit von Millisekunden steuern, indem man ihre Konformation durch Lichtreize bestimmter Wellenlängen ändert. Dies erlaubt eine schnelle und spezifische Kontrolle definierter molekularer Abläufe in biologischen Systemen.
Doktorand Florian Lindner fusionierte die dynamische T3SS-Komponente mit einem Teil eines optogenetischen (lichtabhängigen) Interaktionsschalters, der andere Teil wurde in der bakteriellen Membran verankert. Die Bakterien werden in Kontakt mit den Wirtszellen gebracht und dann an der gewünschten Stelle (und zum gewünschten Zeitpunkt) gezielt mit blauem Licht beleuchtet. Damit lässt sich die Verfügbarkeit der T3SS-Komponenten und damit die Funktion des T3SS an- und ausschalten. Die Forschergruppe nannte ihre Neuentwicklung LITESEC-T3SS: Light-induced translocation of effectors through sequestration of endogenous components of the T3SS.
Anwendung des LITESEC-Systems in der Tumorforschung

Da die Beladung des System (die zu injizierenden Proteine) sehr flexibel ist, kann das System vielseitig eingesetzt werden. In einer Zusammenarbeit mit Prof. Thorsten Stiewe vom Institut für molekulare Onkologie der Philipps-Universität Marburg nutzte die Arbeitsgruppe das neue System, um Tumorzellen in Zellkultur abzutöten (Abb. 2).
Basierend auf diesem Erfolg will die Gruppe diese Technik jetzt sowohl für die Grundlagenforschung (zum Beispiel für ein besseres Verständnis des T3SS), als auch zur Entwicklung weiterer Anwendungen nutzen.














