Die Schlüsselenzyme der biologischen Methanbildung
Forschungsbericht (importiert) 2017 - Max-Planck-Institut für terrestrische Mikrobiologie
Methan ist ein Endprodukt des anaeroben Abbaus von organischem Material und ein starkes Treibhausgas. Etwa die Hälfte der weltweiten Methanemissionen wird von methanogenen Archaeen durchgeführt. Wir interessieren uns für die an der hydrogenotrophen Methanogenese beteiligten Enzyme und berichten über die Kristallstrukturen der Formyl-Methanofuran-Dehydrogenase und des Heterodisulfid-Reduktase/Hydrogenase-Komplexes. Diese Enzymkomplexe sind an den sequentiellen Reaktionen der Ferredoxin-Reduktion und CO2-Reduktion/Fixierung im methanogenen Stoffwechselweg beteiligt.
Methanogenese aus H2 und CO2
In der Natur werden riesige Mengen Methan von methanogenen Archaeen produziert. Wir untersuchen zurzeit in Zusammenarbeit mit Ulrich Ermler vom Max-Planck-Institut für Biophysik, Abteilung Molekulare Membranbiologie, die Struktur und Funktion von methanogenen Enzymen. Im letzten Schritt dieses Stoffwechselweges entstehen Methan und ein Heterodisulfid. Das Heterodisulfid wird durch den Heterodisulfid-Reduktase/Hydrogenase-Komplex (Hdr/Mvh) reduziert, und aufgrund eines Elektronen-bifurkierenden Mechanismus kommt es dabei zusätzlich zu einer Reduktion von Ferredoxin. Das reduzierte Ferredoxin dient der Reduktion und Fixierung von CO2, was durch den Formyl-Methanofuran-Dehydrogenase-Komplex (Fwd) katalysiert wird. Die Kristallstrukturen des Fwd- und Hdr/Mvh-Komplexes zeigten neuartige Cofaktoren und einzigartige Strategien zur Energiekopplung [1, 2].
Der Großteil der methanogenen Archaeen produziert Methan hydrogenotroph aus H2 und CO2. Dabei sind, wie gesagt, spezielle Enzyme und neuartige Coenzyme im Spiel [3]. Hydrogenotrophe Methanogene leiten den Metabolismus typischerweise durch einen CO2-Fixierungsprozess ein. Diese Reaktion wird durch den Fwd-Komplex katalysiert, der CO2 reduziert und nachfolgend an ein Methanofuran-Coenzym fixiert, sodass Formyl-Methanofuran entsteht. Als nächstes wird die Formylgruppe auf Tetrahydromethanopterin (H4MPT) übertragen, was zur Entstehung von Formyl-H4MPT führt. In Folgereaktionen wird die Formylgruppe in eine Methylgruppe umgewandelt, die dann auf Coenzym M (CoM-SH) übertragen wird. Diese Methyl-Transfer-Reaktion ist stark exergon und mit der Translokation von Natriumionen auf der gesamten Membran verknüpft [4]. Die abschließende Reaktion der Methanogenese wird durch Methyl-Coenzym M-Reduktase katalysiert, wodurch Methyl-S-CoM mit Coenzym B (CoB-SH) zu Methan und dem Heterodisulfid CoM-S-S-CoB reduziert wird [5]. Durch die mittels des Hdr/Mvh-Komplex katalysierte Reaktion entstehen erneut CoM-SH und CoB-SH. Innerhalb des Enzymkomplexes bildet die Heterodisulfid-Reduktase einen Komplex mit Hydrogenase, wobei H2 als Elektronendonator dient. Mit den beiden H2-Molekülen als Elektronenquelle reduziert der Hdr/Mvh-Komplex gleichzeitig CoM-S-S-CoB und Ferredoxin. Das reduzierte Ferredoxin dient dann als Elektronendonator für die durch den Fwd-Komplex katalysierte erste Reaktion.
Molekularer Aufbau des Formyl-Methanofuran-Dehydrogenase-Komplex (Fwd)
Zur Aufklärung der molekularen Grundlagen der Heterodisulfid-Reduktion und CO2-Fixierung wurde die Kristallstruktur des Fwd-Komplexes des thermophilen Methanogens Methanothermobacter wolfeii ermittelt [1]. Die Struktur des Fwd-Komplexes ist als Tetramer des Heterohexamers Fwd(ABCDFG)4 mitsamt 46 [4Fe-4S]-Clustern aufgebaut (Abb. 1A).
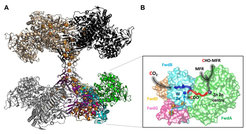
Die meisten biologischen Stoffwechselprozesse zur Fixierung von CO2 beginnen mit dem Einfangen von CO2 in Form einer Carboxygruppe, gefolgt von einer Reduktion. Im Gegensatz dazu wendet der Fwd-Komplex eine andere Strategie an: CO2 wird zunächst zu Formiat reduziert, das nachfolgend, an Methanofuran fixiert, zu Formyl-Methanofuran umgewandelt wird (Abb. 1B). Die zur Formiat-Dehydrogenase homologe Untereinheit FwdB enthält Tungstopterin als aktives Zentrum sowie einen Kanal für den CO2-Eintritt. Zur Reduktion von CO2 zu Formiat werden Elektronen durch die [4Fe-4S]-Cluster auf das Tungstopterin übertragen. Das entstandene Formiat gelangt durch einen 43 Å langen Tunnel zum nächsten aktiven Zentrum.
Bei FwdA handelt es sich um ein Amidohydrolase-Homolog, das die Kondensation von Formiat zu Methanofuran katalysiert. Eine der unerwarteten Besonderheiten des 800 kDa schweren Komplexes ist der aus 46 [4Fe-4S]-Clustern bestehende Elektronendraht (Abb. 1A). Der Elektronendraht könnte als Elektronenrelais fungieren und die vier aktiven Zentren (Tungstopterin) über 206 Å synchronisieren.
Molekularer Aufbau des Heterodisulfid-Reduktase/Hydrogenase-Komplexes (Hdr/Mvh)
Die Kristallstruktur des Hdr/Mvh-Komplexes wurde mithilfe des thermophilen Methanogen Methanothermococcus thermolithotrophicus ermittelt [2]. HdrA stellt vermutlich das katalytische Zentrum eines neuen Mechanismus zur Energiekopplung dar, der sogenannten flavinbasierten Elektronenbifurkation. Dabei werden zur Reduktion von Heterodisulfid beziehungsweise Ferredoxin vier Elektronen in zwei Elektronen geringerer Energie und zwei Elektronen höherer Energie aufgeteilt. Das reduzierte Ferredoxin transferiert hochenergetische Elektronen zur CO2-Reduktion, was durch den zuvor beschriebenen Fwd-Komplex katalysiert wird. Dank der Kristallstruktur des Hdr/Mvh-Komplexes konnten die Elektronentransferwege vom Hydrogenase zum Heterodisulfid-Reduktase-Modul in diesem Enzymkomplex verfolgt werden (Abb. 2).

Die Analyse der Flavin-Umgebung von HdrA liefert Hinweise auf den Mechanismus der Elektronenbifurkation. Da zahlreiche Mikroorganismen HdrA-Homologe in verschiedenen Enzymkomplexen aufweisen, kann die Hdr-Struktur des HdrABC-MvhAGD-Komplexes als Prototyp dieser Proteinfamilie angesehen werden. Darüber hinaus entdeckten wir in der HdrB-Untereinheit eine neue Anordnung von Eisen-Schwefel-Clustern: die sogenannten Nicht-Cuban-[4Fe-4S]-Cluster. Diese neuartigen Eisen-Schwefel-Cluster sind an die konservierten Aminosäurereste der CCG-Motive gebunden. Zwar findet sich das CCG-Motiv auch in rund 2000 anderen Proteinen, seine Struktur und Funktion waren jedoch unbekannt. Anhand der Methode des sogenannten Cryo-Intermediate-Trappings konnte dargestellt werden, wie die Nicht-Cuban-Eisen-Schwefel-Cluster CoM-S-S-CoB reduzieren [2].
Zukünftige Projekte
In den Kristallstrukturen des Fwd-Komplexes zeigte sich der faszinierende, aus 46 [4Fe-4S]-Clustern bestehende Elektronendraht [1]. Zur Erforschung der Funktion des langen Elektronendrahtes wird der Fwd-Komplex jetzt anhand elektrochemischer und spektroskopischer Verfahren untersucht, um die elektronischen Eigenschaften der Eisen-Schwefel-Cluster-Kette aufzuklären. Zur Untersuchung des Mechanismus der Elektronenbifurkation werden weitere, alternative Konformationen des Hdr/Mvh-Komplexes mit einem anderen Abstand zwischen Flavin und den Eisen-Schwefel-Clustern benötigt, die zurzeit hergestellt werden.
Literaturhinweise
Science 354, 114-117 (2016)
Science 357, 699-703 (2017)
Faraday Discussion 198, 37-58 (2017)
Scientific Reports 6: 28226 (2016)

