Die Rolle von RNA-bindenden Proteinen während der pathogenen Entwicklung in Ustilago maydis
Forschungsbericht (importiert) 2004 - Max-Planck-Institut für terrestrische Mikrobiologie
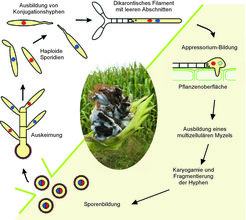
Der Pilz Ustilago maydis ist der Erreger des weitverbreiteten Maisbeulenbrandes. Er gehört derzeit neben dem Humanpathogen Cryptococcus neoformans zu den bestuntersuchten Modellorganismen auf dem Gebiet der pathogenen Basidiomyzeten. Charakteristisch für den Infektionsverlauf von U. maydis sind eine Reihe von morphologischen Veränderungen, die der Pilz durchläuft und die als einfache Entwicklungsprogramme angesehen werden können. Der initiale Schritt der pathogenen Entwicklung ist das Erkennen von kompatiblen pilzlichen Paarungspartnern, bevorzugt auf der Oberfläche von Pflanzengeweben. Die Wahrnehmung spezifischer Pheromone, die von den jeweiligen Partnern sezerniert werden, löst die Bildung von Konjugationshyphen aus, die entlang eines Pheromongradienten gerichtet auf den jeweiligen Paarungspartner zuwachsen (Abb. 1). Nach der Zellfusion wächst das dikaryontische Filament durch unipolares Spitzenwachstum. Da der Zytoplasmabestandteil eine definierte Gesamtlänge nicht übersteigt, zieht der Pilz am distalen Ende leere Abschnitte ein. Auf diese Weise kann er größere Strecken auf der Pflanze zurücklegen, um eine geeignete Eindringstelle zu finden. Ist diese gefunden, dringt der Pilz über Appressorien in die Pflanze ein und bildet ein multizelluläres Myzel. Nach einer Phase der Proliferation in der Pflanze stimuliert das Pilzmyzel die Ausbildung von Tumoren, die aus undifferenzierten Pflanzenzellen bestehen und in denen schwarze Teliosporen heranreifen. Dieses gibt infizierten Pflanzen ein verbranntes Aussehen, daher auch der Name Maisbeulenbrand. Reife Tumore platzen auf und setzen eine Fülle von Teliosporen frei, die hauptsächlich über Windverbreitung einen neuen Wirt finden (Abb. 1).
RNA-bindende Proteine in U. maydis
Aus Modellsystemen höherer Eukaryonten ist bekannt, das RNA-bindende Proteine Schlüsselregulatoren für Entwicklungsprogramme sind. Um die Frage zu beantworten, welche Rolle RNA-bindende Proteine in U. maydis spielen, wurde ein revers genetischer Ansatz gewählt. Über eine bioinformatische Analyse wurden anhand der Genomsequenz von U. maydis mögliche RNA-bindende Proteine dadurch identifiziert, dass sie konservierte RNA-bindende Domänen enthalten. Auf diese Weise konnten bislang 94 mögliche RNA-bindende Proteine identifiziert werden, die sich in vier verschiedene Familien gruppieren lassen (Abb. 2).
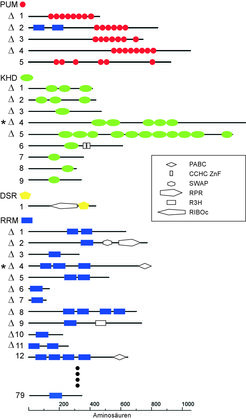
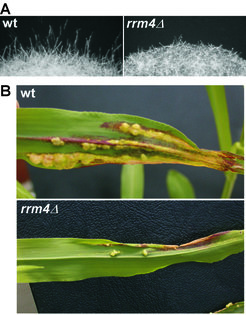
Um deren Funktion zu untersuchen, wurden von ausgewählten Kandidaten über homologe Rekombination entsprechende Deletionsmutanten hergestellt. In einer detaillierten phänotypischen Analyse wurde untersucht, ob diese Stämme in ihrem Wachstumsverhalten, Morphologie, Filamentbildung und/oder Pathogenität gestört waren. Von bislang 25 getesteten Kandidaten führte der Verlust des KHD-Proteins Khd4 oder des RRM-Proteins Rrm4 zu einer abnormalen pathogenen Entwicklung. Da unsere Analyse im Fall von Rrm4 am weitesten fortgeschritten ist, wird auf dieses RNA-bindende Protein im Folgenden besonders eingegangen. Während der phänotypischen Analyse von rrm4 Deletionsmutanten fiel auf, dass diese im Vergleich zu Wildtypstämmen ein vermindertes filamentöses Wachstum aufweisen und vermutlich demzufolge ebenfalls eine verminderte Virulenz zeigen (Abb. 3).
Eine zellbiologische Analyse in einem hierfür besonders geeigneten Laborstamm ergab, dass die Deletion von rrm4 dazu führt, dass ein Großteil der Zellen nicht unipolar, sondern an beiden Zellpolen auswächst. Des Weiteren ist die Fähigkeit gestört, leere Abschnitte einzuziehen, weshalb die Gesamtlänge der Filamente im Gegensatz zum Wildtyp eingeschränkt ist. Um die Funktion von Rrm4 näher zu analysieren, wurde die zelluläre Lokalisation in vivo mithilfe eines Fusionsproteins von Rrm4 und dem grün fluoreszierenden Protein (Gfp) untersucht. Fluoreszenzmikroskopische Aufnahmen von Filamenten zeigten, dass Rrm4-Gfp in Partikeln akkumuliert, die sich bidirektional entlang von definierten Zytoskelett-Bahnen bewegen. Um diese Bahnen näher zu untersuchen, wurden zwei Fusionsproteine co-exprimiert: eine Fusion von Rrm4 mit dem rot fluoreszierenden Protein (Rfp) sowie eine Fusion von α-Tubulin (Tub1) mit Gfp. α-Tubulin ist einer der Grundbausteine von Mikrotubuli. Fluoreszenzmikroskopische Co-Lokalisationsstudien zeigten, dass sich Rrm4-Rfp-Partikel entlang von Tub1-Gfp gefärbten Mikrotubuli bewegen.
Um weitere Aufschlüsse über die Funktionsweise von Rrm4 zu gewinnen, wurden seine verschiedenen Protein-Domänen näher analysiert. Rrm4 weist eine bislang einzigartige Domänenkombination auf. Es besitzt im N-terminalen Bereich drei RRM-Domänen für RNA-Bindung und im C-terminalen Bereich eine PABC-Domäne (Poly[A]-bindendes Protein C terminale Domäne), zuständig für Protein-Bindung. Eine Mutationsanalyse ergab, dass Stämme, die Rrm4-Gfp mit Mutationen in den ersten beiden RRM-Domänen exprimierten, mobile Partikel aufweisen. Allerdings zeigen sie den Deletionsphänotyp, nämlich bipolare Filamente ohne leere Abschnitte. Folglich sind die beiden N-terminal gelegenen RNA-Kontaktregionen RRM1 und RRM2 für die Funktion essentiell. Mutationen in Schlüsselaminosäuren der C-terminalen Protein-Interaktionsdomäne führten dazu, dass sich nur noch vereinzelt unbewegliche Partikel bilden. Da entsprechende Stämme den gleichen Phänotyp wie Deletionsstämme aufweisen, folgerten wir, dass die C-terminale Domäne über Protein-Protein-Wechselwirkungen für die Partikelbildung benötigt wird.
Die bislang erzielten Ergebnisse lassen sich in folgendem Modell zusammenfassen: Die gestörte Filamentbildung in Abwesenheit von Rrm4 zeigt, dass Rrm4-Partikel essenziell für das schnelle, unipolare Wachstum der infektiösen Hyphe sind. Da es sich bei Rrm4 um ein RNA-bindendes Protein handelt, das sich in Form von Partikeln bidirektional entlang von Mikrotubuli bewegt, liegt der Schluss nahe, dass RNA vom Zellkern zu den Zellpolen transportiert wird und sich auf diese Weise ein RNA-Gradient aufbaut (Abb. 4A). Mutiert man die RNA-bindende Domäne, "fahren" Transportpartikel unbeladen. Folglich kann sich kein RNA-Gradient aufbauen und das unipolare Wachstum sowie das Einziehen von leeren Abschnitten ist gestört (Abb. 4B). Wird hingegen die Protein-Interaktionsdomäne mutiert, können gar keine funktionellen RNA-Transportpartikel gebildet werden. Auch in diesem Fall ist das unipolare Wachstum und das Einziehen von leeren Abschnitten gestört (Abb. 4C).

Langstreckentransport von RNA in Eukaryonten
RNA-Transport in Mikroorganismen ist bereits aus der Modellhefe Saccharomyces cerevisiae bekannt: Dort wird die RNA eines Transkriptionsfaktors in die Tochterzelle transportiert, um zu garantieren, dass dieser Faktor spezifisch im Zellkern der Tochterzelle akkumuliert. Diese Transportprozesse verlaufen entlang des Aktinzytoskeletts, das meist für Kurzstreckentransport benötigt wird. Im Gegensatz dazu deuten unsere Ergebnisse daraufhin, dass in U. maydis der RNA-Transport entlang von Mikrotubuli wichtig für das unipolare Spitzenwachstum der infektiösen Hyphe ist. Derartiger Langstreckentransport von RNA ist bisher in Mikroorganismen nicht beschrieben worden. Gut untersuchte Modellsysteme, in denen RNA-Transport entlang von Mikrotubuli ebenfalls wichtig ist, sind die Embryonalentwicklung der Taufliege Drosophila melanogaster oder die synaptische Plastizität in Säugetierneuronen. Die Beteiligung von RNA-Transportprozessen an der pathogenen Entwicklung von U. maydis deutet an, dass solche Prozesse evolutionär ursprünglich in der Entwicklung der Eukaryonten zu sein scheinen.
In Zukunft wollen wir untersuchen, welche RNA-Moleküle für das unipolare Spitzenwachstum transportiert werden müssen, welche Proteinkomponenten Bestandteile der Partikel sind und wie das Zusammenspiel molekularer Motoren diese Partikel bewegt. Von besonderem Interesse ist es, die erzielten Ergebnisse mit denen aus höheren Eukaryonten zu vergleichen, um Unterschiede und Gemeinsamkeiten aufzudecken. Damit liefert die Analyse der RNA-bindenden Proteine in U. maydis nicht nur neue Einblicke in die pathogene Entwicklung, sondern hat darüber hinaus auch das Potenzial, grundlegende Konzepte für andere zelluläre Programme wie Embryonalentwicklung oder neuronale Signalweiterleitung aufzudecken.



