Denitrifikation: Mikrobielle Gemeinschaften und ihre Funktion in der Umwelt
Forschungsbericht (importiert) 2005 - Max-Planck-Institut für terrestrische Mikrobiologie
Einleitung

Denitrifikation (eine spezielle Form der dissimilatorischen Nitratreduktion) ist eine alternative anaerobe Atmung, die ausschließlich von Mikroorganismen zur Energiegewinnung genutzt werden kann. Unter Denitrifikation im eigentlichen Sinne wird die Reduktion oxidierter Stickstoffverbindungen wie Nitrat (NO3-) und Nitrit (NO2-) zu den gasförmigen Produkten Stickstoffmonoxid (NO), Distickstoffoxid (N2O) und elementarem Stickstoff (N2) verstanden. Die Einzelschritte werden durch vier Reduktasen (Nitrat-, Nitrit-, NO- und N2O-Reduktase) katalysiert (Abb. 1).
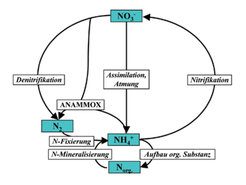
Die Denitrifikation vervollständigt den Stickstoffkreislauf. Einerseits nutzt sie als Substrat Nitrat, das Endprodukt der mikrobiellen Oxidation von Ammoniak (Nitrifikation), andererseits bildet sie mit N2 das Ausgangssubstrat für die Fixierung von Stickstoff (Abb. 2). Daher spielt die Denitrifikation eine entscheidende Rolle in der Aufrechterhaltung eines ausbalancierten globalen Stickstoffhaushalts.
Industriell wird die Denitrifikation in Klärwerken zur Eliminierung von Stickstoff aus Abwässern genutzt. Reduziertes Wachstum landwirtschaftlich genutzter Pflanzen wiederum ist eine unerwünschte Folge der Denitrifikation, da sie den durch Düngung in den Boden eingebrachten Nährstoff Nitrat entfernt und als Gase in die Atmosphäre abgibt. Die dabei gebildeten gasförmigen Produkte tragen entscheidend zum Klimawandel bei. Denitrifikationsprozesse in Böden sind die Hauptquelle von atmosphärischem N2O und verursachen ungefähr 57% der globalen Emission. N2O ist ein starkes Treibhausgas mit einem 300-mal höheren globalen Erwärmungspotenzial als CO2 und trägt zur Erwärmung der Erdatmosphäre und zur Zerstörung der Ozonschicht in der Stratosphäre bei.
Um Denitrifikationsprozesse in der Natur zu verstehen und auf lange Sicht eventuell beeinflussen zu können, ist nicht nur die Kenntnis des Prozesses an sich, sondern auch der Mikroorganismen notwendig, die dafür verantwortlich sind. Das Wissen darüber, um welche Organismen es sich handelt und wie sie in der Umwelt funktionieren, ist jedoch noch sehr begrenzt.
Denitrifizierende Mikroorganismen

Reinkulturen von Denitrifizierern im Labor sind hauptsächlich der Domäne der Bakteria zuzuordnen. Von den kultivierten Stämmen der Bakteria insgesamt sind wiederum ungefähr 50% zur Denitrifikation befähigt. Jedoch wurden auch unter den Archaea und den Pilzen Organismen gefunden, die denitrifizieren können. Diese Fähigkeit ist also weit verbreitet und beschränkt sich nicht auf eine engverwandte Gruppe. Die Gruppe der Denitrifizierer lässt sich also nur über diese spezielle physiologische Fähigkeit definieren, nicht jedoch über ihre stammesgeschichtliche Einordnung.
Reinkulturen zur Untersuchung von Mikroorganismen-Gemeinschaften erfassen jedoch bekanntermaßen nur einen Bruchteil der Diversität von in der Umwelt auftretenden Organismen. In der letzten Dekade wurden molekularbiologische Ansätze, die auf der Detektion von Genen für die vier Reduktasen (Abb. 1) mittels PCR beruhen, entwickelt. Damit lassen sich Denitrifizierer direkt in ihrem Habitat erfassen, ohne sie vorher in Kultur nehmen zu müssen (Abb. 3 und [7]). Die Detektionssysteme basieren zum Beispiel auf den Genen für die Nitritreduktase [4], die als Schlüsselenzym der Denitrifikation gilt, da alle weiteren Produkte des Prozesses gasförmig sind und somit nicht von den Organismen assimiliert werden können.
Diversität von Denitrifizierern in der Umwelt

Molekularbiologische Untersuchungen verschiedenster Habitate haben eine immense Vielfalt von Denitrifizierern aufgezeigt, die weit über die bislang bekannter kultivierter Denitrifizierer hinausgeht. Diese neuartigen Denitrifikanten sind mit den kultivierten Vertretern teilweise nur sehr entfernt verwandt (Abb. 4 und [2, 5, 8]). Dennoch zeigen statistische Analysen, dass die bislang beobachtete Diversität bei weitem noch nicht die gesamte Vielfalt abdeckt [2, 5, 8].
Es ist auch möglich, dass die bisher publizierten Detektionssysteme nur eine begrenzte Vielfalt an Organismen erfassen. Die Entwicklung spezifischer Detektionssysteme ist stets durch die Vielfalt der Gensequenzen begrenzt, die in Datenbanken gespeichert sind. Allerdings werden kontinuierlich weitere Denitrifikationsgene aus Reinkulturen sequenziert. Anhand dieser erweiterten Datenbasis können auch die Detektionssysteme für Denitrifizierer verbessert werden, sodass in Zukunft eine umfangreichere Erfassung möglich sein wird.
Struktur von Denitrifiziererpopulationen
Denitrifiziererpopulationen in Umweltproben werden häufig mittels so genannter ‚fingerprint’-Verfahren wie dem Terminalen Restriktionsfragmentlängen Polymorphismus (T-RFLP) untersucht (Abb. 3). Dafür werden zunächst entsprechende Genfragmente, zum Beispiel der Nitritreduktasegene, durch PCR aus dem Genpool der Umwelt-DNA amplifiziert. Anschließend werden die Amplifikate durch ein Restriktionsenzym fragmentiert und der Länge nach aufgetrennt. Detektiert werden aber nur die terminalen Fragmente, da sie während der PCR mit einem Fluoreszenzfarbstoff markiert wurden. Die Stärke der Signale für einzelne terminale Fragmente lässt sich als relative Häufigkeit eines Genfragments innerhalb einer Population beschreiben, und die Gesamtheit der Signale gibt somit die Vielfalt und Zusammensetzung der Denitrifizierergemeinschaft im Ökosystem wieder. Terminalen Fragmenten in den T-RFLP-Profilen von Umweltproben können durch computergestützte Analysen Fragmente von Datenbanksequenzen zugeordnet werden [9]. T-RFLP-Analysen erlauben die Bearbeitung einer großen Anzahl von Proben und ermöglichen so vergleichende Analysen von Populationen entlang von Umwelt- und Zeitgradienten oder nach Inkubationsversuchen.
In der Umwelt stellen die Denitrifizierer, die bereits aus Kultivierungsversuchen bekannt sind, anteilsmäßig nur eine unbedeutende Gruppe dar. Dominant hingegen sind bislang nicht kultivierbare Mikroorganismen mit neuartigen Denitrifikationsgenen, jedoch unbekannter verwandtschaftlicher Zugehörigkeit [1, 3, 6, 8]. Sie sollen durch neuartige Kultivierungsverfahren, die in letzter Zeit entwickelt wurden und die die nährstoffarme Umwelt dieser Organismen zu imitieren versuchen, in Kultur genommen werden. Für diese Reinkulturen kann dann nicht nur die verwandtschaftliche Zugehörigkeit ermittelt, sondern es können auch physiologische Eigenschaften der Organismen untersucht werden.
Was treibt Struktur und Aktivität von Denitrifizierergemeinschaften?
Verfügbarkeit von Sauerstoff, Nitrat, organischem Kohlenstoff, der pH-Wert und die Temperatur der Umgebung sind Faktoren, die sowohl die Aktivität als auch die Entwicklung von Denitrifizierergemeinschaften steuern. Substratverfügbarkeit wird unter anderem durch Interaktionen mit anderen Organismen im Habitat beeinflusst. Diese Interaktionen sind interessant, aber nur teilweise verstanden, da es sich um komplexe Beziehungen handelt. Ein Beispiel für Interaktionen zwischen Mikroorganismen ist die Verflechtung von Denitrifikation und Nitrifikation über Nitrat als Endprodukt der Nitrifikation beziehungsweise als Ausgangssubstrat der Denitrifikation. So erhöht eine Düngung des Bodens mit steigenden Mengen Ammoniumstickstoff nicht nur die Aktivität, sondern verändert auch die Populationen der Denitrifizierer. Ammoniak wird durch die Aktivität nitrifizierender Bakterien zu Nitrat oxidiert (Abb. 2), das dann als Ausgangssubstrat für die Denitrifikation zur Verfügung steht. Steigende Ammoniakkonzentrationen führen zu einer Erhöhung der Oxidationsraten und über eine vermehrte Bereitstellung von Nitrat zu einer Dominanz von Denitrifizierern, die es effektiv zur Energiegewinnung nutzen können. Die damit verbundene Aktivitätserhöhung führt dann wiederum zu beschleunigtem Nitratverlust und gesteigerter Treibhausgasemission aus dem Boden [1].
Noch komplexer als Interaktionen zwischen Mikroorganismen sind solche zwischen Mikroorganismen und Pflanzen. Man nimmt an, dass mikrobielle Diversität und die Diversität der Vegetation korreliert sind. Das heißt, eine diversere Vegetation bedingt eine komplexere Denitrifizierergemeinschaft. Weiterhin geht man davon aus, dass diversere Gemeinschaften effizienter funktionieren als weniger diverse. Damit würde die Diversität der Planzengemeinschaft sowohl die Zusammensetzung als auch indirekt die Funktion der Denitrifizierergemeinschaft im Boden beeinflussen. Diese Hypothese wurde für Denitrifiziererpopulationen aus Böden von Modellökosystemen, die mit verschiedenen Planzenarten und in verschiedenen Pflanzendiversitätsstufen bepflanzt wurden, getestet. Im Gegensatz zu den ursprünglichen Annahmen beeinflussten jedoch nicht die Diversität des Pflanzenbewuchses, sondern die Artzusammensetzung Struktur und Funktion der Denitrifizierergemeinschaft. Dabei dürften sowohl die Wurzelbiomasse als auch Nährstoffe, die von den Wurzeln an den Boden abgegeben oder frei werden, wenn die Wurzeln absterben, eine entscheidende Rolle spielen. Auch die Konkurrenz um Nährstoffe zwischen Pflanzen und Mikroorganismen könnte wichtig sein. Wurzelbiomasse, Nährstoffart und -menge werden wesentlich durch die Pflanzenart mitbestimmt. Trotz des deutlichen Einflusses der Vegetation war die Zusammensetzung der Denitrifizierergemeinschaft unter verschiedenen Pflanzenmonokulturen jedoch erstaunlich konstant, da die dominierenden Denitrifizierer im Wesentlichen dieselben waren. Nur ein geringerer Teil der Gemeinschaft war charakteristisch für eine Pflanzenart (Abb. 5).

Allerdings kann nur auf einer Analyse der Populationsstruktur basierend nicht geklärt werden, welche Denitrifizierer für die Aktivität im Boden verantwortlich sind. Die Populationsstruktur beschreibt eher das genetische Potenzial einer Gemeinschaft, zu denitrifizieren. Der Prozess selbst wird wahrscheinlich nur von wenigen Mitgliedern der Population ausgeführt. Welche Denitrifizierer jeweils aktiv sind, wird durch die sich stets verändernden Umweltbedingungen bestimmt. Um den Prozess der Denitrifikation in Öksystemen besser zu verstehen, soll sich unsere zukünftige Forschung mit folgenden Fragestellungen befassen: 1) welche Denitrifizierer nehmen eine relevante Funktion in einem Habitat wahr, 2) wodurch ist die Denitrifikation auf molekularer Ebene reguliert und 3) wie wird dieser Prozess von Umweltfaktoren beeinflusst? Die Beantwortung dieser Fragen wird letztendlich die Grundlage sein, Strategien zu entwickeln, um die Denitrifikation zu beeinflussen und somit sowohl die industrielle Nutzung zu optimieren als auch die Treibhausgasproduktion zu minimieren.




