DNA-Spuren bezeugen das Kräftemessen zwischen Viren und Bakterien
Forschungsbericht (importiert) 2010 - Max-Planck-Institut für terrestrische Mikrobiologie
Einführung
Unsere Erde wird dominiert von den kleinsten Lebensformen, den Prokaryoten (Bakterien und Archaeen) mit einer beeindruckenden Population von geschätzten 4-6 x 1030-Zellen. Um eine Vorstellung solch einer immensen Zahl zu bekommen, wurde errechnet, dass die gleiche Anzahl an Pennies gestapelt eine Höhe von einer Billion Lichtjahren erreichen würde [1].
Selbst wenn man bedenkt, dass auf eine Zelle des menschlichen Körpers etwa zehn Bakterienzellen kommen, verblasst die Anzahl der Interaktionen von Bakterien mit Eukaryonten im Vergleich zum Kontakt von Bakterien mit einem anderen Partner, den Viren. So wurde gezeigt, dass Viren über zehnmal häufiger auf der Erde vorkommen als Prokaryoten und komplexe, sowohl nützliche als auch schädliche Interaktionen mit Bakterien etabliert haben. Die Infektion durch ein Virus mit einem lytischen Vermehrungszyklus führt immer zur Lyse, also zum Tod des Wirtsbakteriums.
Die Entwicklung von verschiedenen raffinierten Abwehrmechanismen gegen Viren beweist die anhaltende Gefahr, die von diesen mobilen genetischen Elementen für Bakterien ausgeht. Die wohl bekanntesten Abwehrmechanismen sind die drei Restriktions-Modifikationssysteme, bei denen spezielle Restriktionsenzyme des Bakteriums die eindringende Virus-DNA erkennen und zerschneiden, wohingegen die eigene DNA durch Methylierung modifiziert und geschützt wird. Im Jahr 2007 wurde ein weiteres Abwehrsystem entdeckt, nämlich die antivirale Funktion der CRISPR- Systeme, die uns beim Blick in ein Wirtsgenom einen faszinierenden Einblick in die Geschichte der Virenangriffe bietet.
CRISPR/Cas – ein Immunsystem der Bakterien und Archaeen
CRISPR-Elemente wurden zuerst in den späten 80er-Jahren als sich wiederholende kurze und unterbrochene Genomsequenzen im Modellbakterium Escherichia coli beschrieben [2]. Später wurde erkannt, dass solche Elemente in ungefähr 40% aller Bakterien als auch in fast allen Archaeen vorkommen und dass ein Genom mehrere dieser Elemente enthalten kann. Der Begriff CRISPR wurde eingeführt als Abkürzung für Clustered Regularly Interspaced Short Palindromic Repeats (Cluster von kurzen, palindromischen, sich wiederholenden Sequenzen in gleichmäßigem Abstand), um die Eigenschaften dieser Elemente zu verdeutlichen. CRISPR-Elemente sind gekennzeichnet durch sich wiederholende Sequenzen mit einer Länge zwischen 24 und 48 Basenpaaren (repeats), die von individuellen Sequenzen ähnlicher Länge (spacers) unterbrochen sind (Abb. 1). Die Anzahl der spacer-Einheiten variiert zwischen ein bis zwei und mehreren hundert in verschiedenen CRISPR-Elementen. Selbst sehr ähnliche Bakterienstämme können anhand der An-und Abwesenheit verschiedener CRISPR-Elemente unterschieden werden.
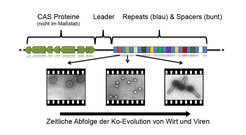
Ein erstes Anzeichen für eine mögliche Funktion dieser mysteriösen CRISPR-Elemente ergab in 2005 die Analyse von Spacersequenzen, die zeigte, dass diese Module in 124 Streptococcus thermophilus Stämmen Homologien zu Sequenzen von Bakteriophagen (77%), Plasmiden (16%), aber auch dem Wirtsgenom (7%) aufwiesen [3]. Ein Durchbruch der Forschung kam im Jahr 2007, als Horvath and Barrango zeigen konnten, dass eine Spacersequenz im CRISPR-Element den Wirt resistent gegen eine Infektion mit einem Virus mit übereinstimmender Sequenz im Genom macht [4]. Die Idee von CRISPR als ein generelles bakterielles Immunsystem war geboren. Weitere Arbeiten konnten zeigen, dass CRISPR auch die Konjugation von Plasmiden, die beispielsweise Antibiotika-Resistenzen in klinischen Isolaten verbreiten, verhindert [5]. Die CRISPR-Funktion wurde auch in Archaeen identifiziert, die sogar eine größere Varibilität an CRISPR Systemen aufweisen [6]. So können hyperthermophile Methanocaldococci über zwanzig verschiedene CRISPR-Elemente aufweisen, die etwa 1% des gesamten Genomes ausmachen.
Mechanismus des CRISPR-Systems
Wie funktioniert ein CRISPR-System als antiviraler Verteidigungsmechanismus? In den letzten Jahren wurden große Fortschritte gemacht, um erste Hinweise zur Funktion von CRISPR-Systemen zu erhalten. Eine erste wichtige Entdeckung war das Vorhandensein von Clustern, bestehend aus konservierten Genen, die oftmals direkt neben den CRISPR-repeats lokalisiert waren. Diese Gene kodieren Proteine, die CAS getauft wurde, was für CRISPR associated sequences steht. Daher werden CRISPR- Elemente oft vereinheitlicht als CRISPR/Cas-Systeme beschrieben. Die Annotation und Unterscheidung der Cas-Proteine erweist sich allerdings als schwierig, da oftmals nur eine geringe Ähnlichkeit auf der Sequenzebene festzustellen ist. Bislang wurden 45 verschiedene CAS-Proteinfamilien postuliert [7], die sich eventuell weiter unterteilen lassen [8]. Mindestens sieben deutlich zu unterscheidende CAS-Protein-Subtypen wurden identifiziert, was die Vielfalt der Funktionen und Wirkungsweisen der CRISPR/CAS-Systeme offenbart.
Der Mechanismus der Immunität durch CRISPR/CAS-Systeme muss zumindest zwei grundlegende Schritte beinhalten, die in der Regel als Adaptation und Interferenz bezeichnet werden:
i.) Adaptation meint die Erkennung des fremden, attackierenden Virusgenoms. Hierbei wird ein Stück der Viren-DNA/RNA (der protospacer) angegriffen und als neuer spacer am 5‘ Ende des Wirts-CRISPR-Clusters eingebaut. Der Mechanismus des spacer-Einbaus ist noch unbekannt. Die Auswahl der protospacer befolgt nur wenige Regeln und scheint ansonsten willkürlich. Zum einen darf ein protospacer nicht auch noch mit Teilen der CRISPR-repeat-Sequenz überlappen; dieser Mechanismus verhindert, dass der spacer nachfolgend im CRISPR-Cluster des Wirtsgenoms nicht selbst als Ziel erkannt und attackiert wird. Zum anderen wird eine kurze Sequenz von zwei bis drei dem protospacer folgenden Basen benötigt, um diesen als solchen auszuweisen.
ii.) Sobald der neue spacer im CRISPR-Cluster eingebaut ist, kann er zur Interferenz, also zur Abwehr neuer Attacken des Viruses, aus dem er ursprünglich stammt, benutzt werden. Dazu wird das CRISPR-Cluster transkribiert und eine lange CRISPR-RNA hergestellt. Diese wird von CAS-Proteinkomplexen in kleinere CRISPR-RNAs (crRNAs) prozessiert. Die crRNAs binden anschließend durch Basenkomplementarität an die angreifende Virus-DNA (oder Virus-RNA, je nach Virustyp) und veranlassen deren Zerstörung durch einen zweiten CAS-Protein- Komplex.
In letzter Zeit wurden Erfolge erzielt, um Details der Funktionsweise der verschiedenen CRISPR/Cas-Systeme auszuarbeiten. Zum Beispiel ist die Generierung von crRNA in verschiedenen Systemen inzwischen schon gut erforscht. Andere Bereiche, wie zum Beispiel der Einbau von spacern, der Mechanismus des Angriffs der Virengenome oder die Evolution und Verbreitung von CRISPR-Systemen, sind noch größtenteils unerforscht und sind im Begriff, ein faszinierendes und neuartiges Forschungsgebiet zu eröffnen.
Eine weitere interessante Fragestellung betrifft den Einbau von Spacersequenzen, die keine Übereinstimmung mit Viren oder Plasmid-DNA haben, sondern derart entworfen werden können, um experimentell beliebige Reporter- oder Wirts-DNA zu attackieren. Damit könnte ein CRISPR/CAS-System eventuell als Mechanismus umgewandelt werden, um gezielt Gene in Bakterien und Archaeen auszuschalten, was den bekannten RNA-Interferenz (RNAi)-Methoden in Eukaryoten ähneln würde. Selbst in den natürlich vorkommenden spacern finden sich Sequenzen des Wirtes und es wird vermutet, dass zum Beispiel CRISPR-Fragmente, Mutationen in CAS-Proteinen oder repeat Sequenzen die Spuren verhinderter Auto-Immunitätsreaktionen darstellen.
Transfer RNA-Gene als Angriffspunkte von Viren
Weitere vermutete Spuren der Ko-Evolution von Viren und Archaeen finden sich in den Genen für tRNAs. Diese kleinen RNAs übersetzen den genetischen Code in die entsprechende Aminosäure im Verlauf der ribosomalen Proteinbiosynthese. Damit sie ihre wichtige Funktion ausüben können, müssen die Moleküle zwei Bedingungen erfüllen. Einerseits müssen sie sich in eine L-förmige Tertiärstruktur falten, um im Ribosom wirken zu können. Andererseits müssen sie bestimmte Sequenz- und Strukurmerkmale aufweisen, die ihre Spezifizität während der Aminosäure-Beladung bestimmen. Zusammengenommen bedingt dies, dass eine Zelle nur sehr wenige Mutationen in solch wichtigen RNAs zulassen kann. Dieses wird in Archaeen von Viren ausgenutzt, die in den meisten Fällen große Teile der tRNA-Gene binden, um sich ins Wirtsgenom zu integrieren. Dabei zerteilt das Virus das tRNA-Gen und rekonstituiert dieses nach erfolgreicher Integration.

Es wurde postuliert, dass diese Integration und das Entfernen von Viren an tRNA- Genen Spuren hinterlässt, die sich als Vorteil in der Evolution erweisen können, falls damit eine Bindestelle des Viruses im tRNA-Gen zerstört wurde [9]. Basis dieser Hypothese ist die Entdeckung von mehreren, unterschiedlich zerstückelten tRNA- Genen in Archaeen (Abb. 2). Ein normales, durchgängiges tRNA-Gen misst im Durchschnitt 76 Basen. Dennoch werden oft tRNA-Gene gefunden, die kurze Intronsequenzen beinhalten. Dabei kann es sogar vorkommen, dass ein tRNA-Gen mehrere Introns gleichzeitig enthält. Eine andere Art und Weise, um tRNA-Gene zu trennen, wurde zuerst in dem Archaeon Nanoarchaeum equitans beschrieben. Hier werden sechs tRNA-Moleküle jeweils von zwei einzeln hergestellten Stücken in trans zusammengefügt [10]. Interessanterweise liegt eine dieser tRNA-Genhälften genau neben einem CRISPR-Cluster. Später wurden tRNA-Gene identifiziert, die auf drei individuelle tRNA-Genfragmente verteilt wurden. Schlussendlich fand man permutiertete RNA-Gene, bei denen die beiden Enden der Gene vertauscht waren.
Die Entstehung und Evolution dieser zerstückelten tRNA Gene wird in den nächsten Jahren in Anbetracht ihrer „molekularen“ Ko-Evolution mit Viren untersucht.

