Molekularer Mechanismus der Eisen-Schwefel-Protein-Biogenese in Eukaryoten
Forschungsbericht (importiert) 2013 - Max-Planck-Institut für terrestrische Mikrobiologie
Die Funktion und Biogenese von Fe/S Proteinen im Überblick
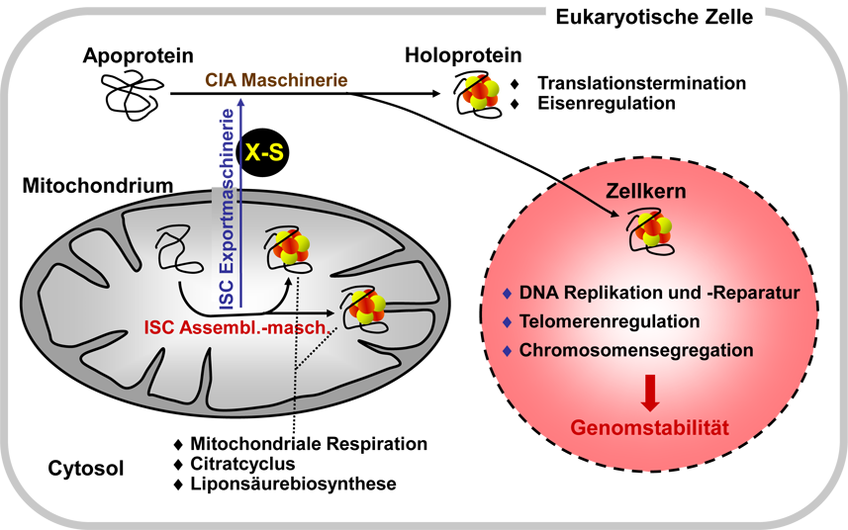
In eukaryotischen Zellen sind Fe/S Proteine in Mitochondrien, Chloroplasten, im Cytosol und im Zellkern lokalisiert. Sie sind an zahlreichen essenziellen Prozessen beteiligt, wie zum Beispiel der mitochondrialen Atmungskette, dem Citratcyclus, der Photosynthese sowie der Biosynthese von Aminosäuren, Nukleinsäuren und Proteinen. Die Beteiligung der Fe/S Proteine an genau diesen Schlüsselprozessen der Zelle erklärt, warum sie für deren Lebensfähigkeit so bedeutend sind. Die häufigsten Fe/S Cluster sind der würfelförmige [4Fe-4S] Cluster und der rhombische [2Fe-2S] Cluster, deren Eisenionen über Cystein- oder Histidinreste an die Polypeptidkette koordiniert sind. Trotz der Einfachheit des Clusteraufbaus erfordert die Biogenese von Fe/S Proteinen in den Mitochondrien und im Cytosol drei komplexe Maschinerien, an denen 30 bekannte Proteine beteiligt sind [1-3] (Abb. 1). In den Mitochondrien wird die iron-sulfur cluster (ISC)-Assemblierungsmaschinerie für die Herstellung aller zellulären Fe/S Proteine eingesetzt. Die cytosolic iron-sulfur protein assembly (CIA) Maschinerie wiederum katalysiert die Reifung cytosolischer und nukleärer Fe/S-Proteine. Dieser Vorgang ist jedoch abhängig von der mitochondrialen ISC Assemblierungsmaschinerie, die eine noch unbekannte, schwefelhaltige Verbindung (X-S in Abb. 1) synthetisiert. Diese Komponente wird mithilfe der ISC Exportmaschinerie aus den Mitochondrien zur CIA Maschinerie transportiert und zur Fe/S Clustersynthese verwendet.
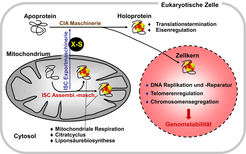
Die ISC und CIA Proteine sind in Eukaryoten, beginnend bei der Hefe bis hin zum Menschen, konserviert. Mehr als die Hälfte dieser Komponenten wird von essenziellen Genen kodiert, das heißt, sie sind unabdingbar für die Lebensfähigkeit eukaryotischer Zellen. Diese Bedeutung ist durch die Funktion einer Reihe von Fe/S Proteinen im Cytosol und Zellkern zu erklären. So sind Fe/S Proteine beispielsweise an der ribosomalen Translation und an verschiedenen Schritten des DNA-Metabolismus einschließlich der DNA-Synthese und Reparatur beteiligt. Letztere Prozesse greifen vor allem auf Fe/S Cluster-haltige DNA-Helikasen zurück, die bei verschiedenen Formen von Krebserkrankungen (z.B. Xeroderma pigmentosum und Fanconi Anämie) mutiert sind und deren Funktionsverlust die Genom-Stabilität beeinträchtigt [4].
Der molekulare Mechanismus der mitochondrialen ISC Assemblierungsmaschinerie
Unsere Forschung der letzten Jahre am Modellorganismus Hefe (Saccharomyces cerevisiae) und an menschlichen Zellkulturen hat zu einem schon recht guten Verständnis des Mechanismus der Fe/S-Proteinbiogenese in Mitochondrien beigetragen. Der Prozess kann in drei Phasen eingeteilt werden und benötigt 17 bekannte Proteine der ISC Assemblierungsmaschinerie (Abb. 2). Die erste Phase umfasst die de novo Synthese eines [2Fe-2S] Clusters. Dazu wird zunächst reduziertes Eisen (Fe2+, rote Kugeln in Abb. 2) durch die Carrier Mrs3-Mrs4 (humanes Mitoferrin) in die mitochondriale Matrix importiert. An diesem Prozess sind die cytosolischen Monothiol-Glutaredoxine Grx3-Grx4 als potenzielle Eisendonoren beteiligt [5]. Die eigentliche Synthese des [2Fe-2S] Clusters beginnt am Cysteindesulfurase-Komplex Nfs1-Isd11, der Schwefel aus Cystein freisetzt und als Persulfid (-SSH) transient an Nfs1 bindet. Der Fe/S Cluster wird dann auf dem Gerüstprotein Isu1 auf chemisch noch unbekanntem Wege synthetisiert. Die Clustersynthese benötigt die Beteiligung von Frataxin (Yfh1 in Hefe), das vermutlich als Eisendonor dient und zusätzlich die Schwefelfreisetzung von Nfs1 stimuliert. Die Reduktion des Cysteinschwefels (S0) zu einem Sulfid (S2-) benötigt die Elektronentransferkette aus NADH, Ferredoxinreduktase (Arh1) und Ferredoxin (Yah1), das wiederum selbst einen [2Fe-2S] Cluster trägt [6].

In der zweiten Phase wird der Isu1-gebundene Fe/S Cluster von Isu1 freigesetzt und auf Apoproteine übertragen. Dies bewerkstelligt ein spezialisiertes, ATP-abhängiges Chaperonsystem, das den Chaperonen der Proteinfaltung stark ähnelt. Dazu gehören die Hsp70 ATPase Ssq1, das DnaJ-ähnliche Co-Chaperon Jac1 und der Nukleotidaustauschfaktor Mge1. Zunächst bindet Jac1 an Isu1 und vermittelt dadurch die spezifische Komplexbildung von Isu1 mit Ssq1. Die resultierende Konformationsänderung führt zur Dissoziation des [2Fe-2S] Clusters und Anbindung an das mitochondriale Monothiol-Glutaredoxin Grx5, das ebenso mit Ssq1 wechselwirkt und so die Clusterübertragung von Isu1 erleichtert. Die bisher erwähnten ISC-Proteine werden als zentrale ISC-Assemblierungsmaschinerie bezeichnet (core ISC assembly machinery), die ausreichend für die Reifung der mitochondrialen [2Fe -2S] Proteine ist. Diese Komponenten sind darüber hinaus auch essenziell für die Bereitstellung der schwefelhaltigen Komponente X-S (Abb. 2, s. unten).
In der dritten Phase wird zum einen der auf Isu1 bzw. Grx5 gebundene [2Fe-2S] Cluster in einen [4Fe-4S] Cluster umgewandelt. Zum anderen wird der [4Fe-4S] Cluster spezifisch in die Polypeptidketten von Zielproteinen integriert. Daran sind mehrere ISC-Proteine, die sogenannten ISC Targetingfaktoren, beteiligt (Abb. 2). Für die Bildung des [4Fe-4S] Clusters wird der Komplex der Isa1, Isa2 und Iba57 Proteine benötigt. Die Isa Proteine binden sowohl Fe als auch einen [2Fe-2S] Cluster, aber ihre genaue molekulare Funktion ist noch nicht geklärt. Der Einbau des [4Fe-4S] Clusters in die verschiedenen Zielproteine wird von zum Teil hochspezifischen ISC Proteinen unterstützt. Allerdings kann der Ausfall eines dieser Proteine manchmal auch von einem der anderen ISC Faktoren kompensiert werden. Die Assemblierung des Atmungskettenkomplexes I erfordert spezifisch die P-loop NTPase Ind1. Die Reifung der Succinatdehydrogenase (SDH oder Komplex II) sowie der Radikal-SAM Fe/S Proteine (Liponsäuresynthase) bedingt die Funktion von Nfu1 und Aim1. Sowohl Nfu1 als auch Ind1 können transient [4Fe-4S] Cluster binden, und man vermutet deshalb, dass sie als Ziel-spezifische Fe/S Cluster-Überträgerproteine fungieren.
Die Rolle der Mitochondrien bei der cytosolischen und nukleären Fe/S Proteinbiogenese
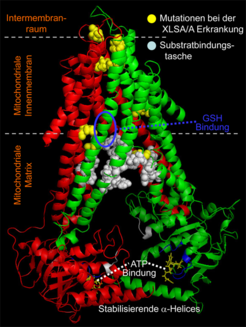
Schon lange ist die bedeutende Rolle der Mitochondrien bei der Reifung von cytosolischen und nukleären Fe/S Proteinen bekannt. Die zentrale Komponente der mitochondrialen ISC Exportmaschinerie ist der ABC-Transporter Atm1 der inneren Membran [7], der die oben erwähnte schwefelhaltige Verbindung X-S ins Cytosol transportiert (Abb. 1 und 2). An diesem Schritt sind ferner das Tripeptid Glutathion (GSH) und die Sulfhydryl-Oxidase Erv1 des Intermembranraums beteiligt, aber ihre genaue Funktion ist unklar. Vor kurzem gelang es, Atm1 der Hefe zu kristallisieren und dessen 3D Struktur aufzuklären (Abb. 3). Das dimere Membranprotein besteht aus zwei ATPase-Domänen und 2x6 Transmembran-Helices, die miteinander verwunden sind und so einen Hohlraum für die Substratbindung bilden [7]. In einer der beiden Atm1 Kristallstrukturen fanden wir GSH in diesem Hohlraum gebunden. Dies lässt vermuten, dass GSH einen Teil des Atm1-Substrates darstellt und im Bereich der Substratbindungstasche des ABC-Transporters angedockt ist. Interessanterweise ist ein GSH-bindender Aminosäurerest im menschlichen Ortholog ABCB7 mutiert und verursacht die Eisenspeicherkrankheit X-chromosomale sideroblastische Anämie und Kleinhirn-Ataxie (XLSA/A; Abb. 3). Weitere mutierte Aminosäurereste bei anderen XLSA/A Patienten befinden sich in der Transmembran-Region des Transporters und könnten den Transport inhibieren. Ein bisher nicht für ABC-Transporter beschriebenes Merkmal ist die C-terminale alpha-Helix, die in Atm1 räumlich vollständig aufgelöst ist. Die beiden Helices des Atm1-Dimers interagieren eng miteinander und fixieren so die beiden ATP-Bindedomänen. Die Folge ist eine Stabilisierung des dimeren Proteins, was funktionell von Bedeutung ist. Die 3D Struktur von Atm1 ist eine wichtige Ausgangsbasis für die Identifizierung seines Substrates.
Die molekulare Funktionsweise der CIA Maschinerie
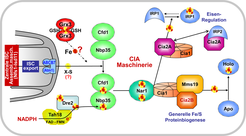
Bisher sind neun CIA Komponenten mit einer Funktion bei der Fe/S Proteinreifung im Cytosol bekannt [3]. Man kann den Prozess in zwei Hauptschritte unterteilen (Abb. 4). Zunächst wird ein [4Fe-4S] Cluster auf dem P-loop NTPase-Komplex Cfd1-Nbp35 synthetisiert, der dort nur transient gebunden bleibt. Die Synthese benötigt die Komponente X-S aus dem Mitochondrium sowie eine Elektronentransportkette, die aus NADPH, dem Flavoprotein Tah18 und dem Fe/S Protein Dre2 besteht [8]. Wofür die Elektronen genau benötigt werden, ist unbekannt. Ebenso ist noch unklar, an welchem Teilschritt die Glutaredoxine Grx3-Grx4 beteiligt sind.
Im zweiten Schritt wird der Fe/S Cluster von Cfd1-Nbp35 abgelöst und spezifisch auf Zielproteine übertragen. Dazu sind die spezifischen CIA Targetingfaktoren Nar1 und der CIA targeting complex nötig, der aus dem WD40 repeat Protein Cia1 sowie Cia2 und Mms19 aufgebaut ist (Abb. 4). Auch die CIA Maschinerie ist von der Hefe bis zum Menschen konserviert. Allerdings besitzen menschliche Zellen zwei Isoformen des Cia2 Proteins. Cia2B als Teil des CIA targeting complex übt die gleiche Rolle wie das Hefe Cia2 bei der Reifung der Mehrheit der cytosolischen und nukleären Fe/S Proteine aus [9]. Cia2A hingegen ist hoch spezifisch für die Assemblierung des iron regulatory protein 1 (IRP1), das eine gut untersuchte Rolle bei der intrazellulären Eisen-Regulation spielt. Ferner bindet Cia2A an IRP2, den Partner des IRP1, obwohl IRP2 keinen Fe/S Cluster trägt. Damit wurde eine bisher unbekannte Verknüpfung zwischen Fe/S Proteinbiogenese und Eisenhomöostase entdeckt, die weiterer Untersuchungen bedarf.
Die Bedeutung der Fe/S Proteinbiogenese für Krankheiten
Die fehlerhafte Biogenese von Fe/S Proteinen kann zu neurodegenerativen, hämatologischen und metabolischen Erkrankungen führen. Mutationen in den humanen Genen von Iba57, Nfu1 und Aim1 (BOLA3) haben schwere mitochondriale Stoffwechselkrankheiten zur Folge, die oft schon in der frühen Kindheit zum Tode führen [10]. Eine weitere bekannte Erkrankung ist die Friedreich Ataxie (Mutationen im Frataxin), die ähnlich wie Mutationen in Isu1 oder Grx5 zu einer Eisenablagerung in den Mitochondrien führt. Die biochemischen Phänotypen der betroffenen Patienten können aus dem geschilderten Mechanismus der Fe/S Proteinbiogenese bereits verlässlich erklärt werden. Jedoch sind mit den Mutationen oft sehr diverse globale Phänotypen verbunden, was generell für mitochondriale Erkrankungen gilt.
Unsere weiteren Untersuchungen widmen sich der Aufklärung der molekularen Grundlagen der Fe/S Proteinbiogenese unter Verwendung zellbiologischer, biochemischer und ultrastruktureller Methoden. Wir versuchen, neue ISC- und CIA- Komponenten zu identifizieren und deren molekulare Funktion aufzuklären. Dies wird auch dazu beitragen, Fe/S Erkrankungen noch besser verstehen zu können.
Danksagung
Gedankt sei der Deutschen Forschungsgemeinschaft (SFB 593, SFB 987 und GRK 1216), der von Behring-Röntgen Stiftung, dem LOEWE Programm des Landes Hessen und der Max-Planck Gesellschaft für die großzügige Förderung der Arbeiten - und all meinen Mitarbeitern für ihre hervorragende Leistung.
![Abb. 2: Modell für den Mechanismus der mitochondrialen Fe/S Proteinbiogenese. Nach der Bildung eines [2Fe-2S] Clusters auf dem Gerüstprotein Isu1 wird der Cluster abgelöst und wenn nötig, in einen [4Fe-4S] Cluster umgewandelt und auf verschiedene Apoproteine übertragen. Die zentrale ISC Assemblierungsmaschinerie wird für alle zellulären Fe/S Proteine benötigt, während die ISC Targetingfaktoren speziell für die Bildung von mitochondrialen [4Fe-4S] Proteinen zuständig sind. Weitere Details siehe Text und Ref. [1, 2]. Pmf, proton motive force; GSH, Glutathion. Abb. 2: Modell für den Mechanismus der mitochondrialen Fe/S Proteinbiogenese. Nach der Bildung eines [2Fe-2S] Clusters auf dem Gerüstprotein Isu1 wird](https://www.mpg.de/11603214/original-1508157089.jpg?t=eyJ3aWR0aCI6ODQ4LCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MDMyMTR9--5faa55138eb3299b82b259720dafacd6cb28cf82)
![Abb. 3: Kristallstruktur des ABC-Transporters Atm1 der mitochondrialen Innenmembran. Grau: Der Hohlraum für die Substratbindung zwischen den beiden Dimeren. Der Bereich der Bindung von Glutathion (GSH) innerhalb des Hohlraums ist mit einer blauen Ellipse markiert. Gelb: Die bei der Eisenspeicherkrankheit XLSA/A mutierten Aminosäurereste im humanen Homolog ABCB7. Weitere Details siehe Text und Ref. [3]. Abb. 3: Kristallstruktur des ABC-Transporters Atm1 der mitochondrialen Innenmembran. Grau: Der Hohlraum für die Substratbindung zwischen den beiden Di](https://www.mpg.de/11603222/original-1508157089.jpg?t=eyJ3aWR0aCI6MzQxLCJmaWxlX2V4dGVuc2lvbiI6ImpwZyIsIm9ial9pZCI6MTE2MDMyMjJ9--35a444901dd593625203dc15890e4bbcca74bfc1)
